我們以 seurat 官方教程為例:
rm(list = ls())
library(Seurat)
# devtools::install_github('satijalab/seurat-data')
library(SeuratData)
library(ggplot2)
library(patchwork)
library(dplyr)
load(file = 'basic.sce.pbmc.Rdata')
DimPlot(pbmc, reduction = 'umap',
label = TRUE, pt.size = 0.5) + NoLegend()
sce=pbmc
如果你不知道 basic.sce.pbmc.Rdata 這個檔如何得到的,麻煩自己去跑一下 視覺化單細胞亞群的標記基因的5個方法,自己 save(pbmc,file = 'basic.sce.pbmc.Rdata') ,我們後面的教程都是依賴於這個 檔哦!
這個官方例子裏面,我們是直接使用了 resolution = 0.5 這樣的方式 :
pbmc <- FindNeighbors(pbmc, dims = 1:10)
pbmc <- FindClusters(pbmc, resolution = 0.5)
實際上這個 resolution 是可以多次偵錯的,比如:
# 參考:https://mp.weixin.qq.com/s/WRhMC3Ojy1GWYfLS_4vSeA
#先執行不同resolution 下的分群
library(Seurat)
library(clustree)
sce <- FindClusters(
object = sce,
resolution = c(seq(.1,1.6,.2))
)
clustree([email protected], prefix = "RNA_snn_res.")
colnames([email protected])
如下所示,可以看到不同的resolution ,分群的變化情況:
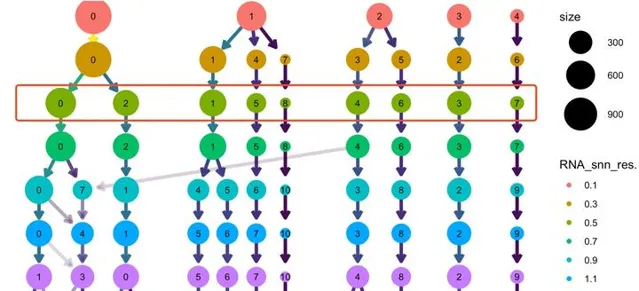
我們是直接使用的 resolution = 0.5 ,僅僅是其中的一個可能性!
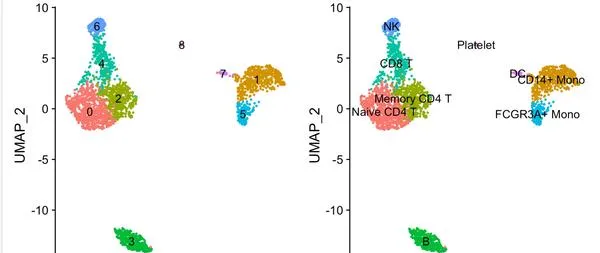
而且根據動態分群的樹,很容易看出來,對應3這個亞群對應的b細胞來說,無論怎麽樣調整參數,它都很難細分亞群了,同樣的還有7這個亞群對應DC,和8這個亞群對應的Platelet也是很難再細分啦。
但是T細胞和monocyte還有進一步細分的可能性!
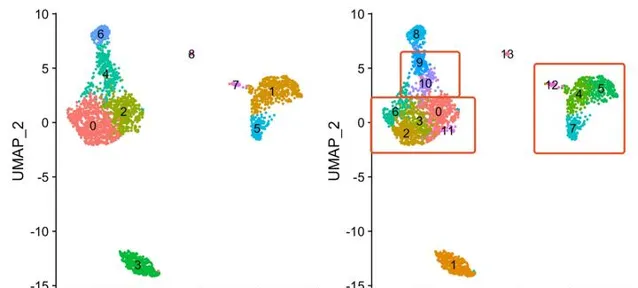
這些視覺化程式碼如下:
p1=DimPlot(sce, reduction = 'umap', group.by = 'RNA_snn_res.0.5',
label = TRUE, pt.size = 0.5) + NoLegend()
p2=DimPlot(pbmc, reduction = 'umap',# group.by = 'seurat_clusters',
label = TRUE, pt.size = 0.5) + NoLegend()
library(patchwork)
p1+p2
p1=DimPlot(sce, reduction = 'umap', group.by = 'RNA_snn_res.0.5',
label = TRUE, pt.size = 0.5) + NoLegend()
p2=DimPlot(sce, reduction = 'umap',group.by = 'RNA_snn_res.1.5',
label = TRUE, pt.size = 0.5) + NoLegend()
library(patchwork)
p1+p2
有分群的可能性,並不代表你一定要進行細分亞群,如果你分群後無法進行準確的生物學描述,那樣細分亞群的意義就不大!
比如前面的CD4的T細胞亞群細分:
load(file = 'sce.cd4.subset.Rdata')
#先執行不同resolution 下的分群
library(Seurat)
library(clustree)
sce <- FindClusters(
object = sce,
resolution = c(seq(.1,1.6,.2))
)
clustree([email protected], prefix = "RNA_snn_res.")
這樣的細分亞群就相互交織:

意義並不是很大











