DNA是細胞遺傳物質的重要載體,基因組復制是所有生物維持遺傳性狀和生理活動的必備過程。復制程式調控如果出現問題,會直接威脅基因組穩定性,是涉及人類衰老以及癌癥等諸多重大疾病的重要因素
【1】
。DNA復制程式的調控主要依賴於復制起始位點和啟動時間的選擇,即DNA復制從哪裏開始,以及每個起始位點何時開始復制。通俗地說,可以將全基因組想象成一個國家的鐵路系統,DNA復制起始位點和復制時間就好比該國境內的所有始發站及發車時間,必須很好的協調。然而對於復制起始位點的研究一直是該領域的難點,存在很大爭議
【2】
。這主要是由於缺乏有效的一致認可的研究復制起點的高通量技術。因為DNA復制程式,即使對於同種細胞系的不同單個細胞來說,也存在細胞間復制起始位點的高度差異,並且絕大部份起始位點的復制效率都非常低(只在1-10%的細胞中使用),這導致復制起始位點的準確檢測異常困難。
因而如何高效準確地辨識復制起點的位置,成為揭示細胞復制機制的關鍵,是目前學科的前沿和熱點。
2021年6月21日,法國居禮研究所
陳春龍
教授、美國麻省醫學院
Nick Rhind
教授、弗羅裏達州立大學
David M. Gilbert
教授 和 加拿大西蒙菲莎大學
John Bechhoefer
教授在
Molecular Cell
上聯合發表標題為
Genome-Wide Mapping of Human DNA Replication by Optical Replication Mapping Supports a Stochastic Model of Eukaryotic Replication
的研究論文。作者
透過Bionano高通量單分子成像技術,將包含熒光標記的復制起點和復制叉軌跡的超長DNA分子(150 kb – 2 Mb,平均長度~300 kb)匹配到人類基因組上,發現了除已知的復制起始位點外的大量新位點組成的復制起始區域。

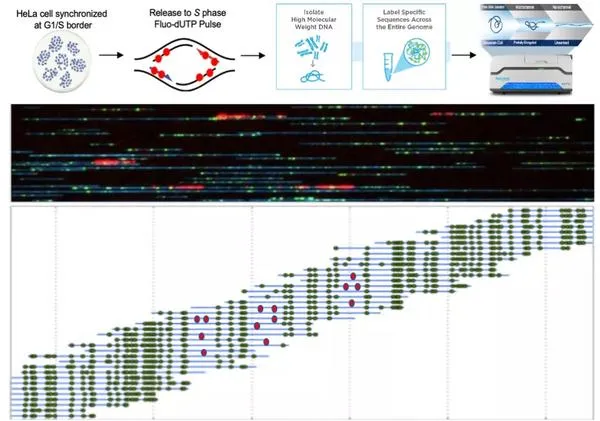
圖1. Optical Replication Mapping(ORM)技術透過高通量單分子成像鑒定復制起始位
Bionano單分子成像是一種全新的基因組光學圖譜技術。Bionano透過全基因組中特定序列(例如限制性內切酶酶切位點)處的熒光基團標記,然後基於奈米微流控芯片技術透過DNA分子在奈米通道(nanochannel)上的運動獲得超長線性單分子(上百kb),最後透過高分辨率顯微鏡成像讀取這些單分子的資訊。透過DNA分子上綠色訊號的相對座標,檢查到的分子可以很容易的匹配到參考序列上,如圖1所示。該技術擁有高通量,低成本,單分子,超長讀長和高匹配準確度等優勢,已廣泛套用於基因組的組裝,基因組結構變異的檢測等領域。
該工作是首次將Bionano技術套用拓展至人類細胞DNA復制領域,作者透過獲得覆蓋率高達整個人類基因組長度的1700倍的單分子數據,能發現使用品頻率低至千分之一的復制起始位點
。同時, 作者發現絕大部份在細胞復制早期檢測到的復制起始位點位於早復制區(一種評估該位點在眾多細胞中平均復制時序被稱為Replication Timing - RT的物理量
【3】
),但仍有部份檢查到的起始位點位於晚復制區域。這打破了傳統學說的嚴格復制時序調控理論,說明了即使在晚復制(細胞群體的平均值)區域中,部份細胞依然可能在此區域選擇起始位點,在S phase的早期復制。
作者透過對細胞進入S期後的不同時間結點的細胞進行分析,根據所檢測到的復制起始位點附近標記的DNA復制標記片段的分布,可以清楚地觀測到隨著S phase的推進,DNA復制從復制起始位的向兩邊推進的漸變過程,這說明了該技術以及其所辨識的復制起點的準確性。同時,除了鑒定復制起始位點,
本研究方法還能檢測復制叉在S期的動態變化。
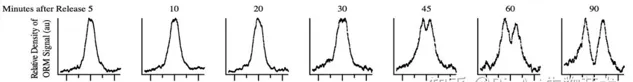
圖2. 隨著細胞復制的推進,ORM技術檢測到的復制訊號從復制起始位點向上下遊延伸
透過進一步的多組學數據分析,作者發現復制起始區域主要富集於基因組上開放的組織蛋白區域,尤其是高表達基因的間隔區。特別是在復制起始區域,揭示了各種可能與復制相關的蛋白例如
pAZ
【4】
(協助DNA復制復合物的載入和起始過程)以及其它開放區域相關的組織蛋白訊號例如
DNAse I,pK4me1
等(常富集於基因表現的強化子或啟動子等區域)的富集。而基因轉錄過程相關的蛋白訊號例如pK79me2,RNA Pol II- phosphoS2等則在復制起始區域的上下遊富集。這些結果與該課題組之前發表的透過測量細胞群體水平的Okazaki fragments(OK-seq)
【5】
獲得的復制起始區的研究結果相吻合。
復制起點富集於基因間隔區很可能是為了避免復制過程與轉錄過程的沖突【6】
。這進一步為ORM技術所發現的復制起始位點的可靠性提供了強有力的生物學佐證。
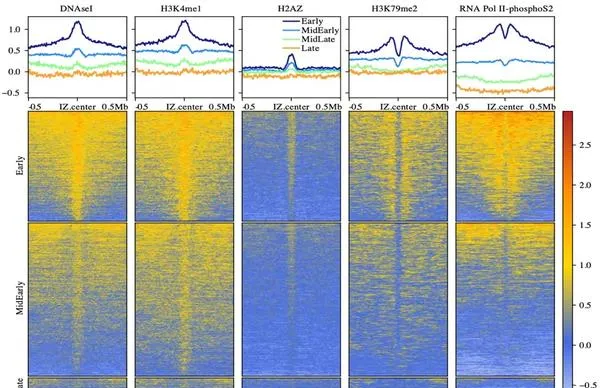
圖3. 代表性分子蛋白訊號在復制起點區域附近的的富集情況
由於本方法的單分子水平特性,能準確的鑒定每個DNA分子(來源於單個細胞)復制起點是否被使用,也可以
準確計算每個復制起點的使用頻率
。透過構建復制起始機率模型
【7】
,利用本工作得到的數據,我們能很好的預測群體水平的復制時序(RT)曲線(與實驗獲得的時序性曲線相關系數高達0.85,遠遠高於其它目前測得的復制起點數據)。這強有力的證明了
復制程式的時序調控是基於復制起始位點的使用頻率
,也就是說是一個以每個位點復制效率的大小為機率的隨機過程。
綜上所述,該研究透過交差學科的融合(細胞與分子生物學,生物資訊學,數理統計,物理模型等),使用創新的基因組學方法,對人類基因組的復制起始位點在單分子水平的鑒定帶來了突破性的進展。
法國居禮研究所的
王緯韜
(Weitao Wang)博士生,美國弗羅裏達州立大學的
Kyle Klein
博士,加拿大西蒙菲莎大學的
Karel Proesmans
博士,以及美國西北大學的
楊紅波
(Hong bo Yang)博士為該文章的共同第一作者,法國居禮研究所
陳春龍
(Chunlong Chen)教授,美國麻省醫學院
Nick Rhind
教授,弗羅裏達州立大學
David M. Gilbert
教授和加拿大西蒙菲莎大學
John Bechhoefer
教授為文章的共同通訊作者。該研究也得到Bionano公司的大力支持。
原文連結:
https://
doi.org/10.1016/j.molce
l.2021.05.024
參考文獻
1. Gnan, S., Liu, Y., Spagnuolo, M. & Chen, C.-L. The impact of transcription-mediated replication stress on genome instability and human disease.
Genome Instab. Dis.
1, 207–234 (2020).
2. Ganier, O., Prorok, P., Akerman, I. & Méchali, M. Metazoan DNA replication origins.
Current Opinion in Cell Biology
58, 134–141 (2019).
3. Vouzas, A. E. & Gilbert, D. M. Mammalian DNA Replication Timing.
Cold Spring Harb. Perspect. Biol.
a040162 (2021). doi:10.1101/cshperspect.a040162
4. Long, H. et al. pA.Z facilitates licensing and activation of early replication origins. Nature 577, 576–581 (2020).
5. Petryk, N. et al. Replication landscape of the human genome.
Nat. Commun.
7, 10208 (2016).
6. Promonet, A. et al. Topoisomerase 1 prevents replication stress at R-loop-enriched transcription termination sites.
Nat. Commun.
11, 3940 (2020).
7. Gindin, Y., Valenzuela, M. S., Aladjem, M. I., Meltzer, P. S. & Bilke, S. A chromatin structure-based model accurately predicts DNA replication timing in human cells.
Mol. Syst. Biol.
10, 722 (2014).
轉載須知
本文著作權歸文章作者所有,歡迎個人轉發分享,未經允許禁止轉載,作者擁有所有法定權利,違者必究。











